Application of chromosomal microarray in diagnosis and treatment of myelodysplastic syndrome
-
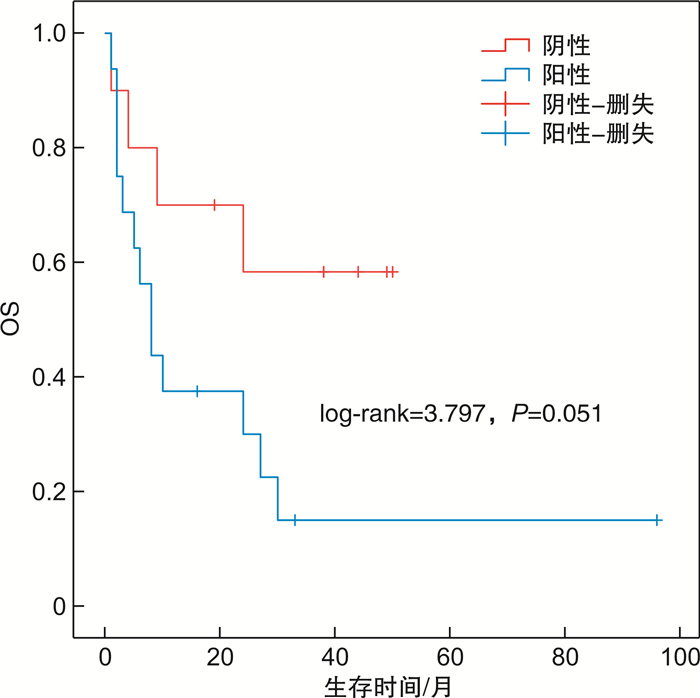
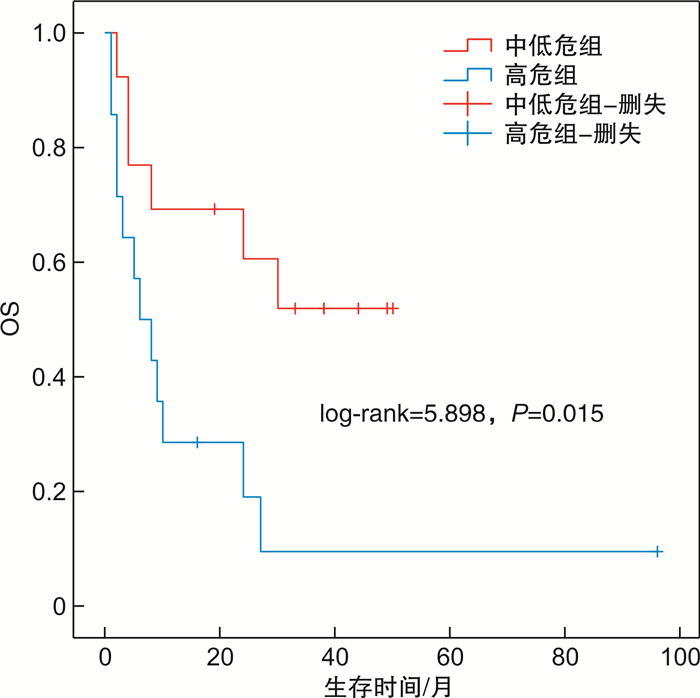
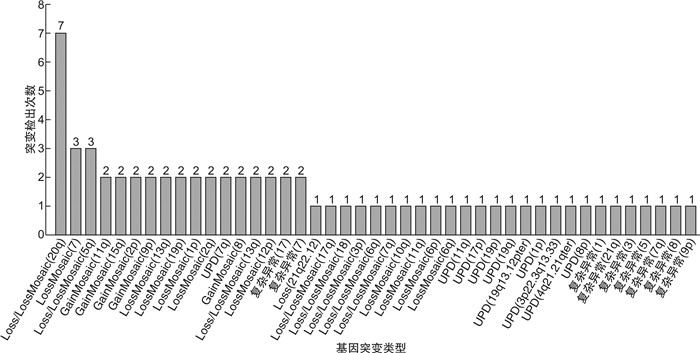
摘要: 目的 探讨染色体微阵列分析(CMA)在指导骨髓增生异常综合征(MDS)的诊断、治疗方式选择及预后判断中的意义。方法 收集2016年3月至2020年6月收治的确诊及疑诊MDS并行CMA检查的36例患者的临床资料,对染色体核型、治疗及预后情况进行回顾性分析。结果 确诊MDS患者27例,其中18例检测到恶性血液疾病相关的染色体改变,占66.67%。其中含Loss、LossMosaic 14例,占77.78%;含GainMosaic 3例,占16.67%;含UPD7例,占38.89%。27例患者中位总生存时间11(1~96)个月。相关性分析显示,患者的临床结局与性别(P< 0.05),是否确诊MDS(P< 0.01),危险度分级(中低危vs高危)(P=0.001)和CMA检测结果(P< 0.01)具有相关性。logistic回归分析提示,CMA检测结果[阴性vs阳性,OR=2.565(95%CI1.059~4.345),P< 0.05]是预测临床结局的独立危险因素。生存曲线分析显示CMA检测为阴性的患者总生存率优于CMA检测为阳性的患者,但两者生存曲线差异无统计学意义(P=0.051)。结论 CMA检测对MDS诊断可以弥补染色体核型分析的不足,提升诊断率,同时可以为MDS患者提供更为准确的生存预测。Abstract: Objective To explore the significance of chromosomal microarray analysis(CMA) in guiding the diagnosis, treatment and prognosis of myelodysplastic syndrome(MDS).Methods The clinical data, karyotype analysis, treatment and prognosis of 36 patients with diagnosed or suspected MDS in the Department of Hematology of our hospital from March 2016 to June 2020 were analyzed retrospectively. All the patients were performed CMA check.Results Among the 27 patients with MDS, 18 cases were detected chromosome changes related to malignant hematological diseases, accounting for 66.67%. There were 14 cases of loss and loss mosaic, accounting for 77.78%; Gainmosaic was found in 3 cases, accounting for 16.67%; 7 cases with UPD, accounting for 38.89%. The median overall survival time of 27 patients was 11(1-96) months. Correlation analysis showed that the clinical outcome of patients was correlated with gender(P< 0.05), diagnosis of MDS(P< 0.01), risk grade(medium and low risk vs high risk)(P=0.001) and CMA test results (P< 0.01). Logistic regression analysis showed that CMA test results (negative vs positive,OR=2.565[95%CI, 1.059-4.345],P< 0.05) were independent risk factors for predicting clinical outcomes. Survival analysis showed that the overall survival rate of patients with negative CMA was better than those with positive CMA, but there was no significant difference between the two survival curves(P< 0.05).Conclusion CMA can make up for the deficiency of chromosome karyotype analysis and improve the diagnosis rate. CMA detection can provide more accurate survival prediction for patients with MDS.
-
-
表 1 36例患者的基线资料
X±S 项目 确诊MDS 疑诊MDS 例数 27 9 年龄/岁 65(31~88) 59(48~79) 性别/例 男 17 3 女 10 6 中性粒细胞/(×109·L-1) 1.58(0.02~22.69) 1.93(0.69~9.51) 血红蛋白/(g·L-1) 74(34~117) 83(54~119) 血小板/(×109·L-1) 49(6~511) 71(13~1162) 随访时间/月 11(1~50) 19(9~34) 表 2 确诊MDS恶性血液系统疾病相关改变检出情况
编号 恶性血液疾病相关的获得性改变 CNV多态性改变 临床意义暂不明确 正常人常见UPD改变 染色体检查结果 1 [LossMosaic(7、11q)]和[GainMosaic(8)] Gain(14q) Loss(22q11.23、Xq26.2q26.3) UPD(3p) 46,XY,-7,+8,del(11)(q21)[5]/46,XY[6] 2 UPD(1p) 无 Gain(1q) 无 46,XY[20] 3 Loss/LossMosaic(3p、5q、7q、20q) 无 无 Gain(14q) 44,XY,-3,del(5)(q13),der(7) dic(3;7)(p21;q22) der(3) del(3)(p11.2p14) inv(3)(p26p21),der(20) del(20)(q11) dic(20;22)(p13;p11.2),-22[19]/46,XY[1] 4 [Loss/LossMosaic(6q、18、20q)]和复杂异常[(7q、9p、17、21q)] 无 UPD(Xp) UPD(Xq11.1q13.1、Xq13.2q21.1、Xq22.3q23) 47,XX,del(7)(q22q23),del(9)(p13),-17,-18,del(20)(q11),+mar1,+mar2,+mar3,+mar4[17]/46,XX[3] 5 [Loss(20q)]和[UPD(11q)] Gain(14q) UPD(1q) 无 46,XY,del(20)(q11)[20] 6 [Loss/LossMosaic(13q、17q)][UPD(19p)] 无 UPD(3q) 无 46,XY[20] 7 Loss(21q22.12) Gain(14q) UPD(7q22.3q31.1、Xp21.1p11.4) UPD(3p21.31p21.1、Xq11.1q21.1) 46,XX[20] 8 UPD(4q21.21qter) 无 无 无 46,XY[20] 表 3 临床资料间的相关性分析
项目 临床结局 CMA检测阳性 MC阳性 相关系数 P 相关系数 P 相关系数 P 年龄 0.054 0.753 0.076 0.659 0.002 0.992 性别 0.335 0.046 0.125 0.468 0.068 0.692 确诊MDS 0.577 < 0.001 0.516 0.001 0.513 0.001 危险度分级 0.538 0.001 0.628 < 0.001 0.307 0.068 是否化疗 0.058 0.738 0.026 0.881 0.097 0.572 MC结果 0.276 0.104 0.438 0.008 — — CMA检测结果 0.559 < 0.001 — — 0.438 0.008 表 4 临床结局单因素logistic回归分析
项目 OR(95%CI) P 年龄(< 60岁vs ≥60岁) 1.600(0.310~8.247) 0.574 性别(男vs女) 3.250(0.611~17.283) 0.167 危险度分级(中低危组vs高危组) 7.000(1.098~44.607) 0.039 CMA检查结果(阴性vs阳性) 6.500(1.094~38.633) 0.040 染色体核型检查结果(阴性vs阳性) - 0.999 是否化疗(否vs是) 0.318(0.059~1.705) 0.181 -
[1] 中华医学会血液学分会. 骨髓增生异常综合征中国诊断与治疗指南(2019年版)[J]. 中华血液学杂志, 2019, 40(2): 89-97.
[2] 朱雨, 何广胜. 世界卫生组织2016年骨髓增殖性肿瘤及骨髓增生异常综合征/骨髓增殖性肿瘤分类更新解读[J]. 中国实用内科杂志, 2016, 36(8): 658-661, 667. https://www.cnki.com.cn/Article/CJFDTOTAL-SYNK201608011.htm
[3] Byrd JC, Mrózek K, Dodge RK, et al. Pretreatment cytogenetic abnormalities are predictive of induction success, cumulative incidence of relapse, and overall survival in adult patients with de novo acute myeloid leukemia: results from Cancer and Leukemia Group B(CALGB 8461)[J]. Blood, 2002, 100(13): 4325-4336. doi: 10.1182/blood-2002-03-0772
[4] Grimwade D, Walker H, Oliver F, et al. The importance of diagnostic cytogenetics on outcome in AML: analysis of 1, 612 patients entered into the MRC AML 10 trial. The Medical Research Council Adult and Children's Leukaemia Working Parties[J]. Blood, 1998, 92(7): 2322-2333. doi: 10.1182/blood.V92.7.2322
[5] Greenberg P, Cox C, LeBeau MM, et al. International scoring system for evaluating prognosis in myelodysplastic syndromes[J]. Blood, 1997, 89(6): 2079-2088. doi: 10.1182/blood.V89.6.2079
[6] Haase D, Germing U, Schanz J, et al. New insights into the prognostic impact of the karyotype in MDS and correlation with subtypes: evidence from a core dataset of 2124 patients[J]. Blood, 2007, 110(13): 4385-4395. doi: 10.1182/blood-2007-03-082404
[7] Solé F, Luño E, Sanzo C, et al. Identification of novel cytogenetic markers with prognostic significance in a series of 968 patients with primary myelodysplastic syndromes[J]. Haematologica, 2005, 90(9): 1168-1178.
[8] Pozdnyakova O, Miron PM, Tang G, et al. Cytogenetic abnormalities in a series of 1, 029 patients with primary myelodysplastic syndromes: a report from the US with a focus on some undefined single chromosomal abnormalities[J]. Cancer, 2008, 113(12): 3331-3340. doi: 10.1002/cncr.23977
[9] Mohamedali AM, Gäken J, Ahmed M, et al. High concordance of genomic and cytogenetic aberrations between peripheral blood and bone marrow in myelodysplastic syndrome(MDS)[J]. Leukemia, 2015, 29(9): 1928-1938. doi: 10.1038/leu.2015.110
[10] Stevens-Kroef MJ, van den Berg E, Olde Weghuis D, et al. Identification of prognostic relevant chromosomal abnormalities in chronic lymphocytic leukemia using microarray-based genomic profiling[J]. Mol Cytogenet, 2014, 7(1): 3. doi: 10.1186/1755-8166-7-3
[11] Maciejewski JP, Tiu RV, O'Keefe C. Application of array-based whole genome scanning technologies as a cytogenetic tool in haematological malignancies[J]. Br J Haematol, 2009, 146(5): 479-488. doi: 10.1111/j.1365-2141.2009.07757.x
[12] Gondek LP, Tiu R, O'Keefe CL, et al. Chromosomal lesions and uniparental disomy detected by SNP arrays in MDS, MDS/MPD, and MDS-derived AML[J]. Blood, 2008, 111(3): 1534-1542. doi: 10.1182/blood-2007-05-092304
[13] 国家卫生健康委员会临床检验中心产前筛查与诊断室间质量评价专家委员会. 染色体微阵列分析实验室技术要求专家共识[J]. 中华检验医学杂志, 2019, 42(9): 745-751. doi: 10.3760/cma.j.issn.1009-9158.2019.09.006
[14] 周庆兵, 胡晓梅, 刘峰, 等. 骨髓增生异常综合征的遗传学检测研究进展[J]. 中国实验血液学杂志, 2011, 19(6): 1536-1540. https://www.cnki.com.cn/Article/CJFDTOTAL-XYSY201106042.htm
[15] Xiao X, He X, Li Q, et al. Single-Nucleotide Polymorphism Array Technique Generating Valuable Risk-Stratification Information for Patients With Myelodysplastic Syndromes[J]. Front Oncol, 2020, 10: 962.
-



 下载:
下载: